
摘要
耐药结核病是2035年实现全球终结结核病流行目标的巨大障碍。世界卫生组织 (World Health Organization, WHO) 倡导采用快速分子药物敏感性检测技术进行耐药结核病早期诊断,而覆盖全面可靠的耐药检测靶标是提高分子药物敏感性检测技术可靠性的关键。WHO于2023年11月发布《结核分枝杆菌耐药相关基因突变目录 (第2版)》,目的是基于更广泛的全球数据汇总形成更为全面准确的耐药相关基因突变目录,为开发与完善基于测序或其他方法的新型分子药物敏感性检测技术提供支持。本文对第2版目录相较于第1版目录在分析流程与耐药突变等内容的更新进行了详细的解读,并对未来目录完善的方向进行了展望。
【关键词】分枝杆菌,结核;抗药性,细菌;微生物敏感性试验;基因, 突变
2022年,全球估算新发结核病患者1060万例,其中,耐多药/利福平耐药结核病患者45万例。推广应用快速准确的药物敏感性试验 (简称“药敏试验”) 技术对于耐药结核病患者早期诊断具有重要临床意义。近些年,随着快速分子药敏检测技术的广泛应用,利福平耐药筛查率得到显著提高。2012年,全球仅7%的病原学阳性结核病患者接受利福平耐药性检测;到2022年,病原学阳性结核病患者利福平耐药筛查率增加到73%,并且在此期间,开展耐多药/利福平耐药治疗的患者从77 321例增加到175 650例,充分表明实验室诊断在指导结核病治疗决策中的核心作用。世界卫生组织 (World Health Organization, WHO) 建议对所有结核病患者进行利福平和异烟肼耐药性检测,并且对耐多药/利福平耐药患者进行氟喹诺酮类 (fluoroquinolones, FQs) 药物耐药性检测。已有商品化的分子药敏检测试剂盒可以完成上述药物耐药性的检测,但目前尚缺乏能够快速诊断抗结核新药和再利用药物耐药性的分子检测技术。WHO在2023年7月发布《应用新一代靶向测序技术检测耐药结核病:快速通告,2023》,推荐了几种基于靶向测序的耐药结核病分子诊断技术。新一代靶向测序技术可以通过对临床标本直接检测,实现对十几种抗结核药物耐药性的快速判定,但基于目前对部分二线抗结核药物尤其是新药的耐药分子机制仍然知之甚少,从而限制了靶向测序耐药检测技术的检测效能。
2021年WHO发布第1版《结核分枝杆菌耐药相关基因突变目录》(以下简称《目录》),第1版《目录》包含高质量、全面的表型耐药相关基因突变列表及其置信度分级,但纳入菌株的地理区域代表性较差,且有关新药和再利用药物耐药相关基因突变的数据非常有限。因此,WHO于2023年修订并发布《结核分枝杆菌耐药相关基因突变目录 (第2版)》。新版《目录》包含约52 000株结核分枝杆菌的全基因组测序数据及表型药敏试验结果,纳入分析菌株数量较第1版增加14 000株,同时,修订的《目录》创建了分析方法,药物覆盖范围也更加全面,包括所有一线抗结核药物 (利福平、异烟肼、乙胺丁醇、吡嗪酰胺),以及A组 (左氧氟沙星、莫西沙星、贝达喹啉、利奈唑胺)、B组 (氯法齐明) 和C组 (德拉马尼、阿米卡星、链霉素、乙硫异烟胺、丙硫异烟胺) 二线药物。笔者重点从第1、2版《目录》差异入手,对《结核分枝杆菌耐药相关基因突变目录 (第2版)》进行解读。
一、优化分析流程
第2版《目录》相较于第1版,提供了更加集中、高效、开源、可持续的分析流程,以减少未来《目录》更新的工作量。在第1版《目录》中,所有表型/基因型药敏试验数据和所有计算过程,均基于访问受限的学术高性能计算集群。在第2版《目录》中,所有数据和计算过程都转移到WHO监督的云服务器中,构建基于开源关系型数据库管理系统的样本标准数据库 (GenPhenSQL),并将后续生物信息学处理及耐药相关性统计分级的分析流程存储在Github中 (https://github.com/GTB-tbsequencing/mutation-catalogue-2023)。整个分析流程可移植到任何云服务器上运行,实现了数据的规范化管理与标准化分析。
(一) 数据采集
第1版《目录》涵盖的菌株相关分析数据集主要来自出版物、CRyPTIC联盟,以及各个国家为响应WHO全球结核病规划贡献的呼吁而直接提交的数据集。第2版《目录》在此基础上实现了与国家生物技术信息中心 (National Center for Biotechnology Information, NCBI) 或欧洲核苷酸档案馆数据库 (European Nucleotide Archive, ENA) 的同步更新,可以获取更多公开数据。因此,第2版《目录》纳入菌株数量更多,67个国家贡献菌株数量大于5株,耐药菌株所占比例也有显著提升,尤其是新药及再利用药物耐药菌株。
(二) 表型药敏数据
第2版《目录》对表型药敏试验结果的可信度进行了更细致分层。其根据WHO对不同药敏试验方法的认可程度,将表型药敏试验数据分为8个层级,前3个层级为使用WHO推荐试验方法得到的数据集 (WHO数据集)。第1层级包括根据最新WHO推荐的关键浓度所得到的基于固体培养或者MGIT液体培养方法的药敏试验数据,以及以1 mg/L作为利福平关键浓度和以0.4 mg/L作为异烟肼关键浓度获得的显微镜观察药敏试验 (MODS) 数据;第2层级为基于低于之前WHO推荐的关键浓度,但高于当前WHO推荐的关键浓度获得的莫西沙星药敏试验数据;第3层级包括根据基于之前的WHO推荐的关键浓度所得到的基于固体培养或者MGIT、BACTECTM 460液体培养方法的药敏试验数据;第4层级包括未标注所采用WHO推荐关键浓度版本的基于固体培养或者MGIT BACTECTM 460液体培养方法获得的药敏试验数据;第5~8层级为其他表型药敏试验方法获得的数据,包括基于微量肉汤稀释法的最小抑菌浓度 (minimum inhibitory concentration, MIC) 药敏试验等。这种分层方式能够创建更为标准化的数据集,为后续分析奠定基础。
(三) 全基因组数据分析方法
第2版《目录》对全基因组测序数据分析方法进行了更新,采用Freebayes识别小于15 bp的变异,并加入delly识别大片段缺失,可获得更为全面和准确的变异列表。保留等位基因频率≥75% (第1版为90%) 的变异进行后续关联分析,同时,对等位基因频率阈值下调到25%进行额外评估,有助于探究异质性耐药对药敏试验结果的影响。除此之外,第2版《目录》采用人类基因组变异协会命名法对变异进行了规范化注释。
(四) 基因突变耐药关联分级方法
第2版《目录》补充完善基因突变与耐药相关性分级方法,扩充了候选耐药基因,并规范了其上游/增强子序列的范围。第1版《目录》仅包括了49个候选耐药基因,第2版《目录》扩充到68个,增加的基因包括参与甘油代谢的glpK,以及参与类固醇和有机酸代谢的Rv1129c等,部分基因从第1版《目录》候选基因中移除,例如,fprA及aftB从阿米卡星的第2层级耐药候选基因中移除。第1版《目录》中候选耐药突变的范围包括各个耐药候选基因及其上游100 bp的范围,第2版《目录》中对每个基因的上游区域都进行了定义。第2版《目录》进一步完善“附加分级规则”。例如,修订了功能缺失突变的定义,将框内突变从功能缺失突变中移除;将交叉耐药性加入附加规则中,Rv0678和pepQ突变会导致贝达喹啉和氯法齐明交叉耐药,fabG1和inhA突变会导致乙硫异烟胺和异烟肼交叉耐药 (表1)。第2版《目录》修改了第4/5组 (可能与敏感相关的突变) 算法,仅将在WHO数据集中,突变单独发生时的阳性预测值95%置信区间上限小于10%的突变列为第4/5组,将不满足上述要求的同义突变也归为第4组。

二、更新耐药基因突变目录
(一) 基因突变及其分级列表
第2版《目录》共有246个1组 (与耐药相关) 突变,其中,包括53个第1版的2组突变及18个第1版的3组突变,另有8个突变未出现在第1版《目录》。第2版《目录》共有1122个2组 (暂定与耐药相关) 突变,包括107个第1版3组突变,126个未在第1版《目录》中出现的突变。需要注意的是,第1版《目录》中有13个1组突变下调到了2/3组。此外,第1版《目录》的106个2组突变下调到第3/4组,主要是框内突变 (表2)。新药和再利用药物耐药相关基因突变显著增多,其中,贝达喹啉、氯法齐明在第1版《目录》中未找到满足1/2组要求的突变,第2版《目录》中贝达喹啉增加了5个1组突变和81个2组突变,氯法齐明增加了2个1组突变及56个2组突变。对于利奈唑胺,第1版《目录》有1个1组突变,第2版《目录》在此基础上增加了7个2组突变。对于德拉马尼,第1版《目录》有1个2组突变,第2版《目录》在此基础上又增加了23个2组突变。除此之外,第2版《目录》对异烟肼和莫西沙星的耐药相关突变进行了高/低水平耐药的标注。
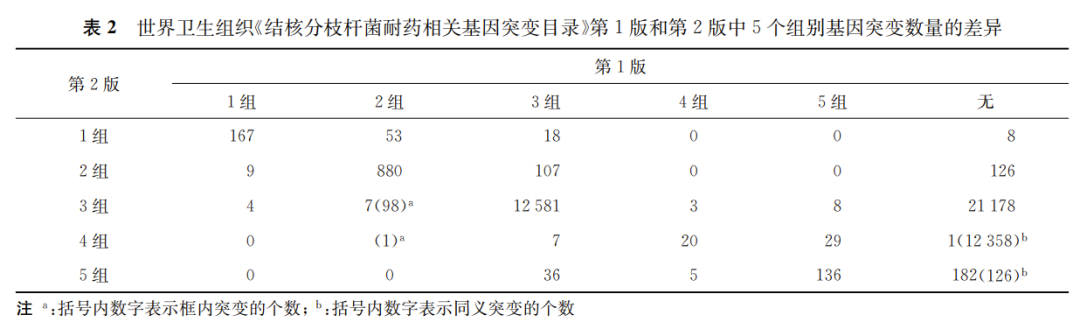
(二) 基因型耐药预测性能
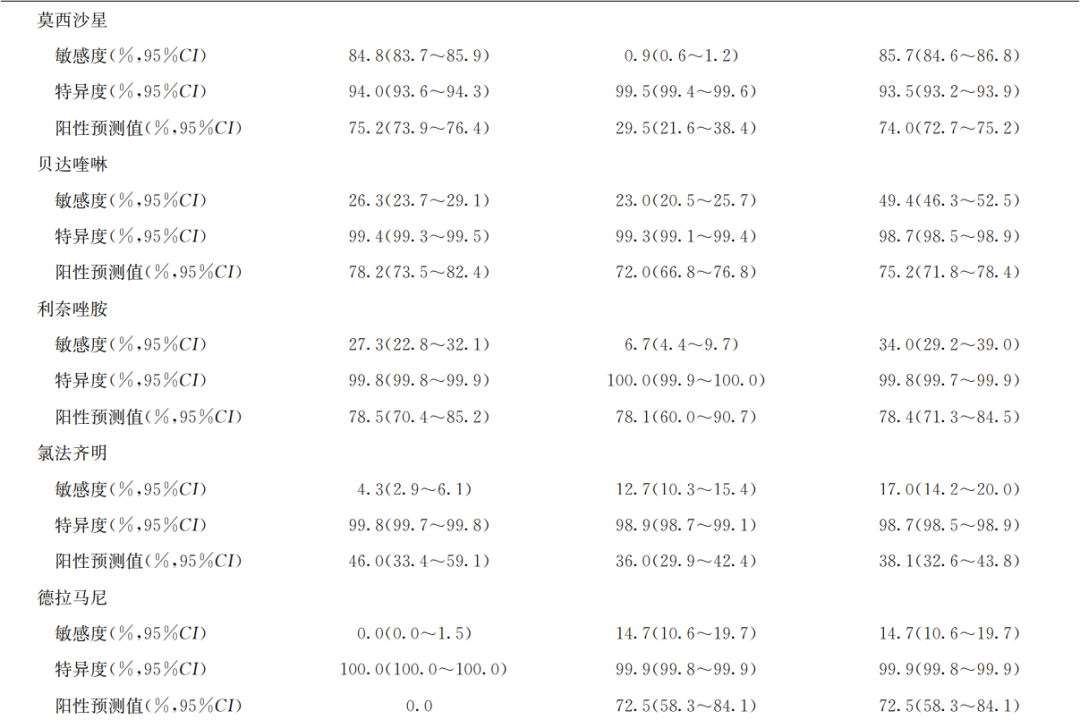
第2版《目录》对大多数药物基因型耐药预测效能值与第1版《目录》较为接近,1组和2组突变组合预测的敏感度≥70%,特异度≥90% (表3)。2组突变对利福平、异烟肼、乙胺丁醇及氟喹诺酮类药物的耐药预测效能无明显影响,但对于吡嗪酰胺和乙硫异烟胺,2组突变能够明显增加其耐药预测效能。第2版《目录》对新药和再利用药物的基因型耐药预测效能均明显提升,但耐药检测敏感度仍不足50%,对新药和再利用药物的耐药机制还有待深入研究。



三、未来《目录》研究展望
(一) 完善数据集
通过收集更多菌株全基因组测序和抗结核药物MIC数据,用以探究基因突变与耐药水平高低的相关性。《结核病整合指南模块3:诊断:结核病快速诊断》中表明,异烟肼和莫西沙星的耐药水平对治疗效果具有明显影响,但目前关于不同耐药基因突变类型所造成的耐药程度差异还有待进一步证实。例如,是否所有katG突变都会赋予高水平异烟肼耐药性,对于较罕见的氟喹诺酮类药物耐药突变,需要更多的MIC值去确定其耐药水平的高低。一些突变仅造成MIC值的小幅升高,如果仅升高到临界浓度附近,则很难对其进行耐药与敏感的精准判读。需要进一步增加纳入分析数据的代表性,降低数据偏倚;同时,还要覆盖更多谱系菌株以探究谱系对耐药性的影响,并收集更多既有耐药菌株又有敏感菌株的数据集,以降低对耐药性的高估。
(二) 优化分析流程
关于测序数据生物信息学分析流程,应进一步加入大片段插入 (如IS6110) 分析,研究其对药物耐药性的影响,并从核苷酸水平探究核苷酸突变对耐药性的影响,包括导致同一氨基酸突变的不同核苷酸突变对耐药性的影响[。现有生物信息学分析流程主要针对二代高通量测序数据,未来应考虑构建同时适用于三代测序数据的分析流程。对于基因突变耐药关联分级方法,一方面要继续优化“附加分级规则”,例如rpoB T427A虽然在利福平耐药性决定区 (RRDR) 中,但可能不导致利福平耐药。还需要继续更新候选耐药基因,例如,dprE2 (Rv3791) 可能是德拉马尼和普托马尼的潜在耐药相关基因。未来建议通过加入回归等其他的分级方法,对补偿性及上位性突变进行深入研究与分类。
四、结语
目前,商品化分子药敏检测试剂盒已广泛应用于临床耐药结核病诊断。随着测序技术的普及,以高通量测序技术为基础的新一代靶向测序试剂盒也将逐步应用于耐药结核病诊断。这些技术可以在短时间内获得多种药物的耐药性信息,进而指导治疗决策。分子药敏检测技术可靠性和实用性的提高取决于对抗结核药物耐药机制研究的不断进步,《目录》可为分子药敏检测靶标选择提供标准化参考。但当前《目录》的算法仅考虑药物候选基因突变与耐药的相关性,未能对新的耐药相关基因进行挖掘。基于《目录》所产生的海量数据,未来在机器学习及神经网络算法的支持下,可以尝试从基因组学出发,对新的耐药相关基因进行探究。此外,《目录》在实用性上也有待进一步提升。《目录》中1组和2组突变位点与目前商品化分子检测试剂盒的检测靶点存在一定的差异 (如ahpC突变位点),且《目录》中包含的耐药突变位点数目众多,但商品化分子药敏检测所能覆盖的检测靶点有限,需要筛选出发生频率高、与高水平耐药相关等特征的突变位点作为商品化分子药敏检测试剂盒的检测靶点。因此,建议未来《目录》对目前已有的分子药敏检测技术覆盖的检测靶点进行评估,并且对耐药相关位点进行靶点推荐性排序。除此之外,还需要考虑《目录》在更新过程中的一致性。例如,第2版《目录》与第1版《目录》在突变的命名上有较大的改变,不利于对两版《目录》进行对照。建议对每次更新过程中新增和移除的耐药相关突变进行额外评估,为更新的必要性提供更多的证据支撑。未来,相信《目录》在更多数据集的支持下,将能够进一步提高通过耐药基因突变预测抗结核药物耐药性的效能,尤其是对新药和再利用药物耐药性的预测。
利益冲突 所有作者均声明不存在利益冲突
文章来源:中国防痨杂志,2024, 46(3): 260-266.
doi:10.19982/j.issn.1000-6621.20230450
基金项目:国家重点研发计划 (2022YFC2305204)
作者:裴少君1, 欧喜超2
作者单位:1北京大学公共卫生学院全球卫生学系,北京 100191;2中国疾病预防控制中心传染病溯源预警与智能决策全国重点实验室结核病预防控制中心,北京 102206
通信作者:欧喜超,Email:ouxc@chinacdc.cn
作者贡献 裴少君:采集数据、起草文章;欧喜超:起草文章、对文章的知识性内容作批评性审阅和指导
本文转载自订阅号「中国防痨杂志期刊社」(ID:zgfl1934)
原链接戳:标准解读 | 世界卫生组织《结核分枝杆菌耐药相关基因突变目录(第2版)》解读
本文完
责编:Jerry


