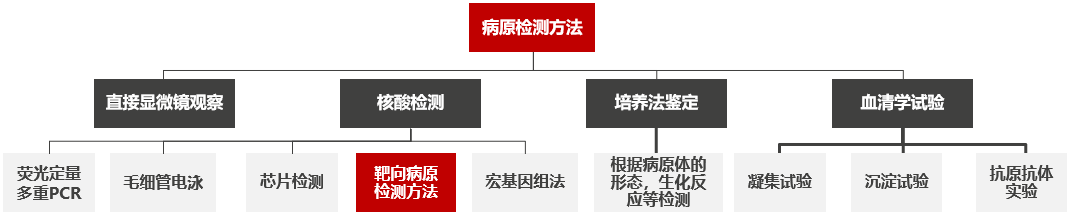
今天与大家分享「基于纳米孔测序tNGS技术诊断肺炎病原体的评估」,下呼吸道感染发病率高、病死率高,是全球第四大死亡原因,在NGS之前,我们有很多传统的病原检测方法,包括直接显微镜观察、核酸检测、培养法鉴定、血清学试验,其中传统分子多重PCR检测简单快速,但检测范围小,需要先假设-后实验-再验证;缺少大量临床重要靶标,而「病原靶向测序(tNGS)」属于核酸水平检测,将样本中目标病原体的核酸进行富集,通过高通量测序平台,得到富集核酸的序列信息,再通过生物信息学手段鉴定样本中的病原体,比传统方法的敏感性更高、比PCR方法的靶标更全、比mNGS技术更便宜经济,整个流程更快,适用于病原学的初筛,剩下的疑难/危重/特殊/罕见病原体交给其它方法如宏基因测序mNGS。
现有病原学诊断手段未能满足临床需求,存在何种挑战?
确定病原体及其耐药性是呼吸道感染治疗的关键,然而还面临多种挑战。
挑战1:二代测序mNGS病原和耐药性鉴定准确性不足。我们之所以选择研究三代测序,实际上是为了将耐药领域纳入其中。二代测序读长短,病原区分度差,耐药性鉴定困难,纳米孔测序是一种单分子、实时测序的新一代方法,读长长,病原区分度佳,耐药性鉴定可行。
挑战2:基于二代测序的mNGS的耐药诊断不成熟。我们在分子药敏、耐药预测方面花费了大量精力,希望通过常见药敏表型和耐药预测为患者提供一些帮助,目前还存在3大难点:关键耐药基因不明确(需建立我国呼吸道病原耐药基因库、遗传标志物库);基因-细菌关联有困难(需构建物种特异耐药谱库、「标志物组合-物种」关联大数据模型);基因型与表型不吻合(还需要基于机器学习的耐药标志物预测策略、大规模临床验证评估、智能耐药分析软件)。
挑战3:mNGS方法价格昂贵。而tNGS是通过超多重PCR与高通量测序的结合,对样本中几十种至几百种已知病原微生物及其耐药基因进行检测。对低浓度的病原微生物的检测,特别是其耐药基因检测,与mNGS相比,具有病原谱范围明确、测序成本低等优势。也就是说,tNGS流程、报告周期更短,费用更低,能够减低成本、覆盖核心病原。
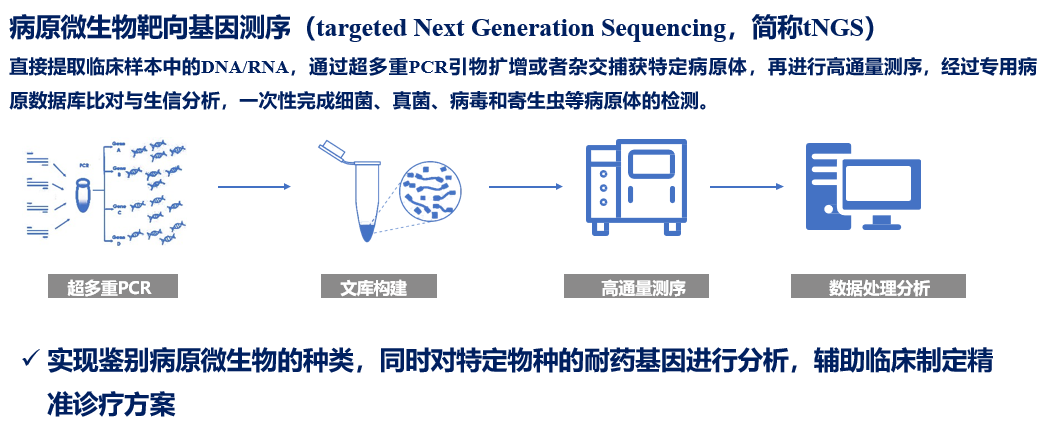
图:tNGS检测原理,超多重PCR/杂交捕获 + NGS = tNGS
tNGS和mNGS的区别?
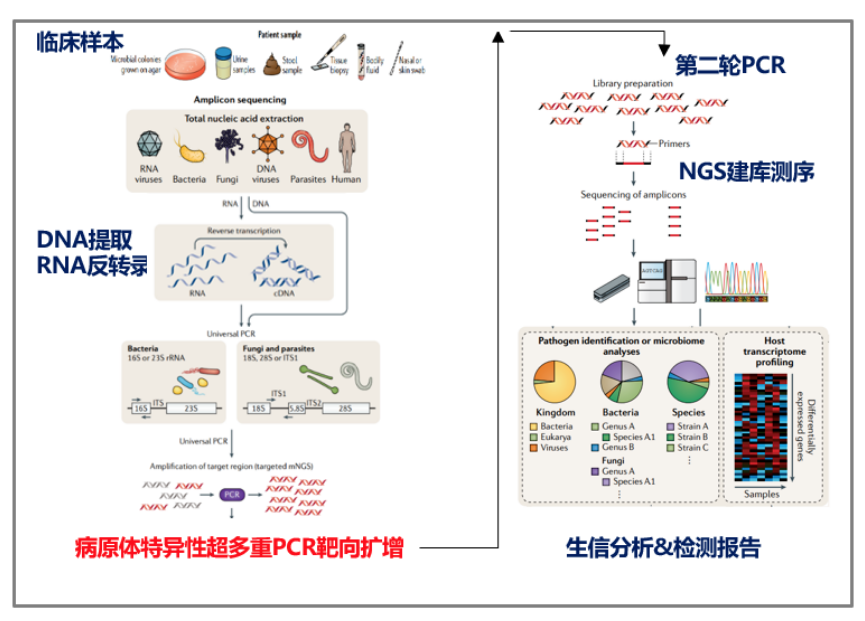
我们可以看看tNGS与mNGS的技术路线对比,tNGS实验步骤:1、样本总核酸提取(包括人的核酸,致病菌,定植菌核酸等);2、富集目标病原体的核酸;3、通过高通量测序平台,获得靶向捕获后核酸的序列信息;4、数据分析。mNGS实验步骤:1、样本总核酸提取(包括人的核酸,致病菌,定植菌核酸等);2、总核酸片段化,末端修复,加A,链接测序接头,文库扩增;3、上机测序,合并;4、数据分析(20Mreads,分析需要计算资源多)。
因此,tNGS和mNGS在技术上最大的区别在于,tNGS存在目标病原体的富集。有了目标病原体的富集,对「去除人源化」方面的要求会更低一些,我们能够花费更少的精力和成本去实现去人源化,并将主要目标放在「富集」方面。从下方的对比图可见,正是由于测序平台的差异,临床诊断方法学也大致分为了靶向和非靶向两大类,两者的差异在于测序前是否针对性的富集某些特点的基因序列,或者是随机放大全部的核酸分子。
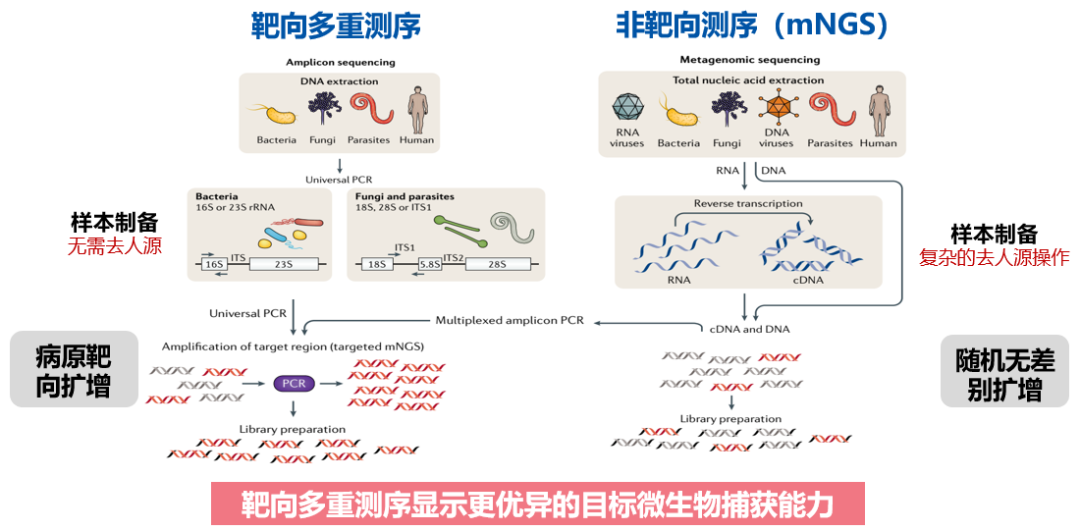
在二代测序的病原菌诊断方法(对所有核酸进行随机无差别扩增)上,靶向测序的技术由于集中对关心的病原菌序列进行扩增、测序分析,期间无需人源化,对目标病原体的检测能力可能会更强;而非靶向测序(mNGS)需要复杂的去人源操作,随机无差别扩增。
国际上有很多文献探讨「病原核酸富集」,它有利于低载量核酸检测、具有信噪比优势,基于富集后的tNGS技术基本不检测人源核酸,降低了「噪音」;减少数据量也就降低了检测成本;当靶向富集,放大靶向信号时能够提升灵敏度。
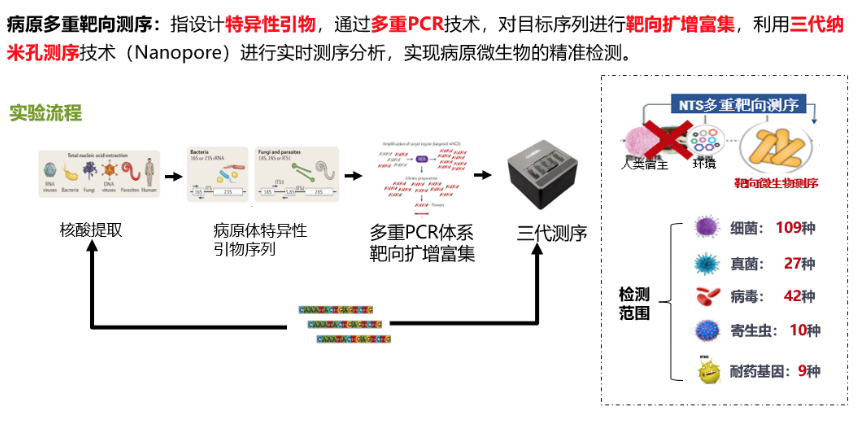
tNGS技术识别病原体的效果如何?优势和劣势在哪?
做tNGS检测有两个策略,第一个是采用多重扩增法。我们采用的是188种病原体+9种主要耐药基因,认为效果不错,引物的干扰较少超多重PCR扩增并非无限扩增病原体,当引物过多时,需要考虑引物之间的干扰。
我们将188种病原体+9种主要耐药基因扩增后,利用三代纳米孔测序技术进行实时测序分析,一些企业还采用探针捕获方法测序,下表为李培教授提供,探针捕获技术:扩增产物较长,更适合于大型病毒及高遗传多样性病毒的分析;PCR扩增子:扩增后的基因产物长度较短(150-200 bp),更适合于预期多态性较低的场景。我个人认为探针捕获法测序已经是界于传统tNGS和mNGS之间的方法,能覆盖的病原体更多,但检测周期也会更长。
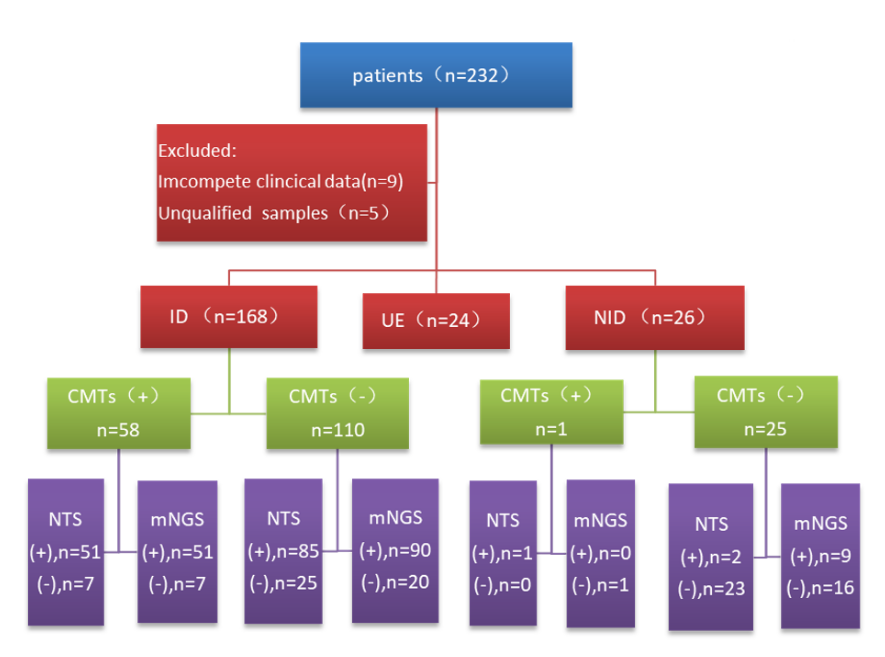
我们tNGS初步尝试所采用的方法是建立纳米孔tNGS的规范化流程,188种病原体+9大类耐药基因,浙江省大约10个中心参与临床研究,收集了232例临床符合肺炎诊断的患者,对这些患者进行了肺泡灌洗,肺泡灌洗液分为三份:一份用于院内传统的病原学检测,包括涂片培养和一些常规的PCR检测;第二份使用院内的mNGS二代测序方法;第三部分使用NTS,即基于三代测序的tNGS测序。在入组的232例患者中,我们发现有168例是真正在住院时被诊断肺部感染的;有26例最初符合肺炎诊断标准,但实际上并非肺炎,例如机化性肺炎或间质性肺炎;还有24例我们认为最终诊断并不明确,所以没有再分析结果。
在168例最终诊断肺部感染的患者中,传统方法明确了58例病原体、111例没有明确病原体;在58例传统方法明确病原体病例中,NTS明确了51例,mNGS也明确了51例;在传统方法阴性病例中,NTS明确了85例,mNGS明确了90例。
在我们的研究数据中,mNGS最主要的优势是厌氧菌检测方面(肺脓肿、吸入性肺炎);从整体上看,对于最终被临床判定为有意义的细菌、真菌和病毒性病原体的检测,传统方法敏感率较低,而mNGS二代测序和NTS基本能够满足要求,在发现真菌性病原体如曲霉时,mNGS相较于NTS要好一些,目前曲霉在NTS中有些假阴性,mNGS阳性率更高。
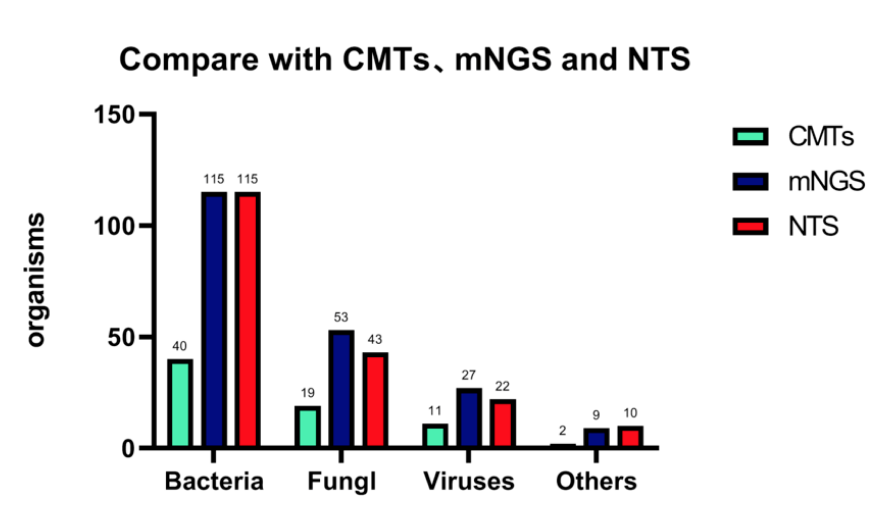
图:主要病原检测效力
这是基于病原学热图的分析:
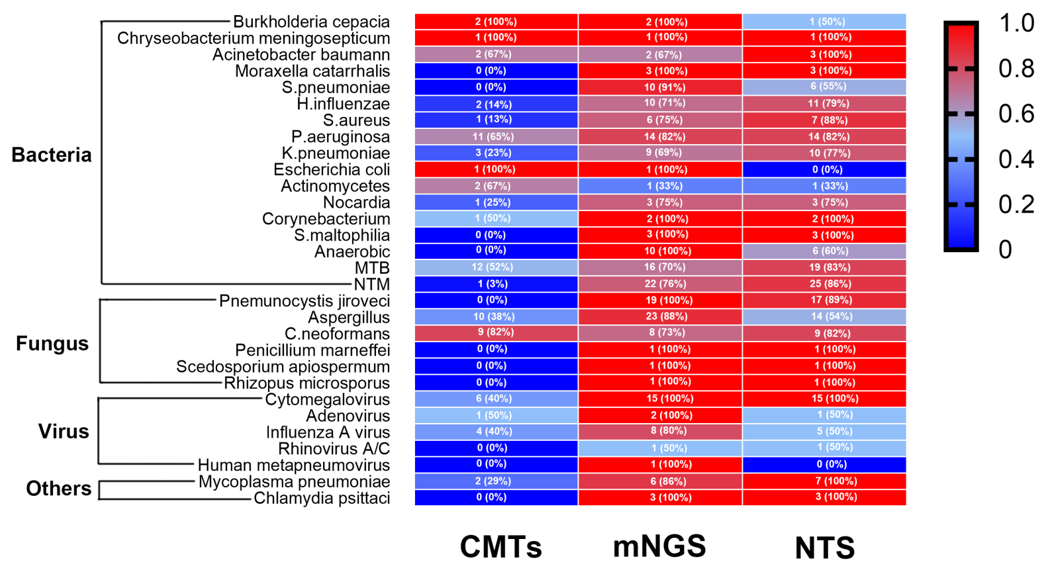
可以看到,在劣势方面,23例患者通过mNGS检测到曲霉,NTS仅检测到14例,RNA病毒中,流感病毒通过mNGS检测到了8例,而NTS仅检测到5例。另外,主要优势在哪里?在TB的检测中,NTS相较于传统的mNGS更具优势。但从统计学分析角度来看,不同病原检测效力没有统计学差异。
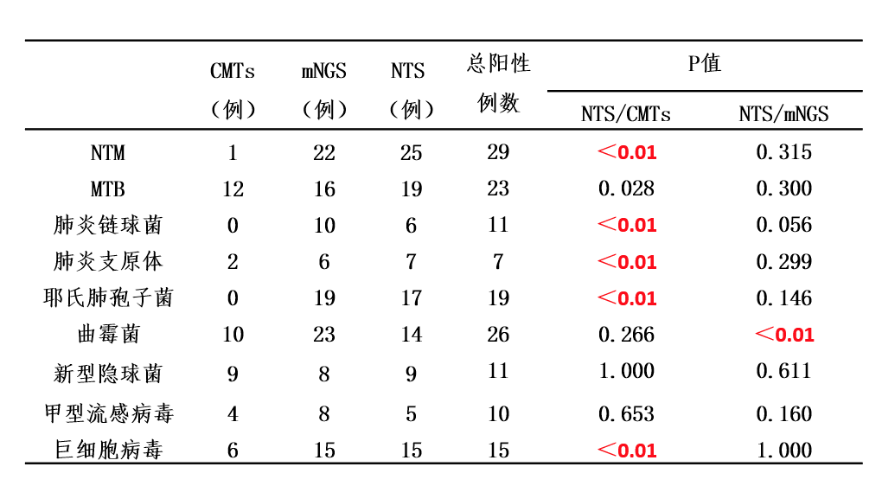
图:9种主要病原检测效力
在混合性病原体检测中,尤其是被诊断为肺脓肿和吸入性肺炎的患者身上,mNGS技术对厌氧菌的检测能力优于NTS,因为NTS所纳入的厌氧菌明显较少。
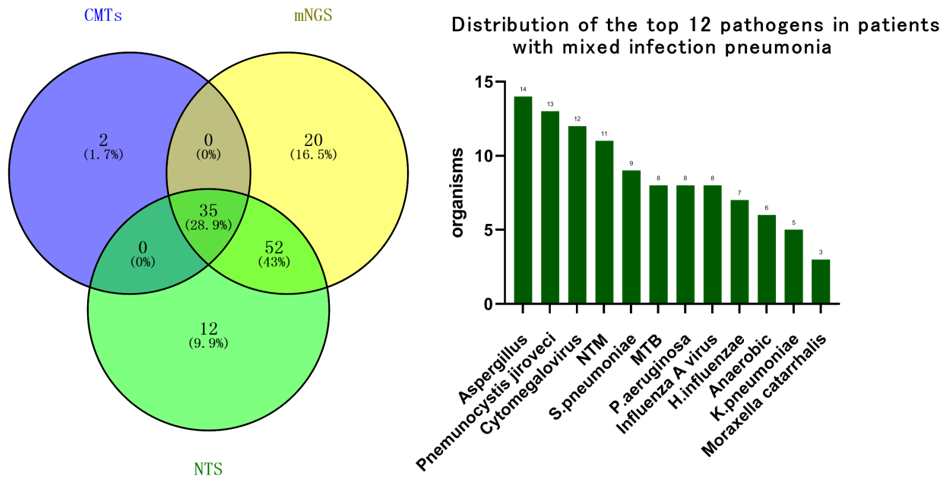
图:混合感染的病原诊断效力
整体上NTS特异性优于mNGS、敏感性略低于mNGS,考虑到成本,我相信它仍可作为临床一个重要的病原检测方法,特别是对于常见的肺部感染患者,因为它的成本可能仅为mNGS的1/3,以上是我们当时对「病原学诊断」的尝试。
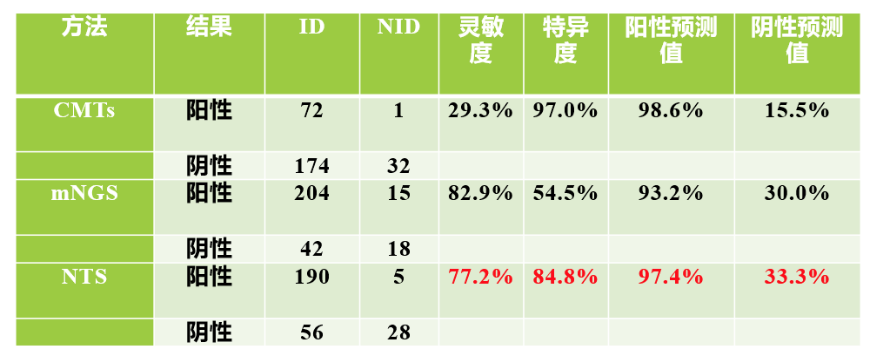
图:CMTs、mNGS和NTS的诊断性能
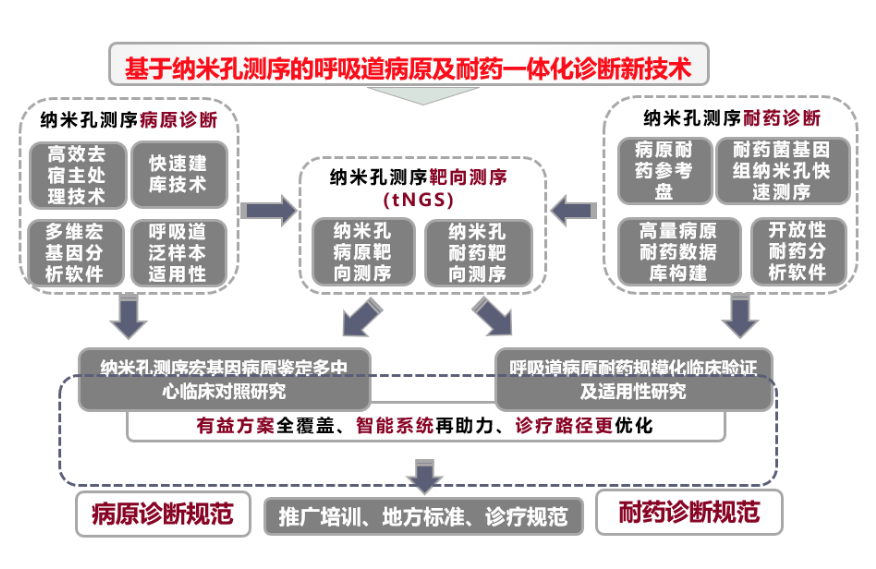
在tNGS技术评估中还设计了另一个模块,即「耐药诊断」
研究入组了另外10家医院的400多例的患者,为什么做耐药诊断?对于一些院内感染,如果仅明确病原体而无法明确耐药性的话,可能临床用药仍无法做到非常精准。因此,传统病原鉴定与药敏试验正在发生变革,当前病原学鉴定以及药敏试验流程复杂且耗时,无需培养的快速病原鉴定与药敏试验方法正在兴起。
国际上提出在高通量测序平台下进行耐药预测是可行的,《JCM》曾发表综述指出全基因组测序(WGS)和宏基因组学技术(mNGS)可用于细菌耐药性预测;《Nature Reviews Genetics》发表综述指出mNGS可用于病原体鉴定,耐药/毒力基因检测。
目前已经完成了约4000例左右的各种临床来源的耐药细菌全基因组测序。拥有了这些细菌的基因组数据、耐药表型后交给机器去学习。研究者们发表了一篇文章:机器学习已广泛用于分子药敏预测研究,在全基因组水平上,预测如肠杆菌目、金葡、结核分枝杆菌等细菌的药敏已取得较好的效果。

相关研究还包括DeepAMR利用突变位点预测结核分枝杆菌对利福平、异烟肼、吡嗪酰胺和乙胺丁醇四种一线药物的单药药敏和预测多耐药、泛耐药结核的模型AUC均在0.95以上;王辉等利用k-mer对国内的金黄色葡萄球菌对克林霉素、头孢西丁、红霉素、复方新诺明、庆大霉素等的MIC进行预测,分类一致率均大于90%;Nguyen M等利用XGBoost模型预测非伤寒沙门氏菌15种药物的MIC,整体准确率(±2倍稀释)大于95%。
我们希望基于AI学习+大数据分析+自动化耐药分析系统(包括数据库构建、机器学习的标志物筛选和预测、分析体系构建、临床评估和AI软件研发),通过纳米孔测序mNGS进行呼吸道感染病原耐药预测、诊断的尝试,这是本项目的第二个研究内容,也是本研究的关键。
目前仅完成了单中心分析,来自单中心44例样本的耐药鉴定发现,纳米孔测序耐药鉴定总体药敏报告率为77.3%,显著高于培养阳性率(55.9%)。原因在于许多病原体经过培养后呈阴性,导致无法进行药敏。从报告率角度来看,由于诊断效率较高,药敏报告率也相对较高。
还发现纳米孔测序预测的耐药结果与金标准培养药敏的分类一致性为95.4%,细菌性病原体主要是院内感染常见的鲍曼、铜绿、肺克,与传统方法进行药敏和耐药检测的符合率接近94%;该中心发现的真菌大部分是念珠菌,无论念珠菌是否感染,预测的耐药结果和培养的药敏分类一致性达到100%。因此,这似乎是一份较好的药敏报告,能为临床医生提供较好的参考依据。
小结
此次我与大家简短分享了肺部感染病原诊断的发展,目前,分子诊断大放异彩,包括多联检、tNGS、mNGS、数字PCR;可以利用mNGS数据进行耐药数据分析、鉴别诊断、预后分析、肿瘤识别;各类技术也在百花齐放,如ddPCR、T2、抗原抗体检测等。总体来说,我个人认为tNGS技术能替代一部分mNGS诊断,因为其效率、速度更快,成本也更低,基于这种方法产生的药敏诊断结果对临床也是重要参考。
参考文献 (可上下滑动浏览)
[1] Marotz CA,et al. Microbiome. 2018 Feb 27;6(1):42.
[2] Burbano HA, et al. Science. 2010;328(5979):723-725.
[3] Vasala A, Hytönen VP, Laitinen OH.Front Cell Infect Microbiol. 2020 Jul 15;10:308.
[4] Su M, Satola SW, Read TD. J Clin Microbiol. 2019 Feb 27;57(3):e01405-18. Chiu CY, Miller SA. Nat Rev Genet. 2019 Jun;20(6):341-355.
[5] Machine Learning for Antimicrobial Resistance Prediction: Current Practice, Limitations, and Clinical Perspective. Clin Microbiol Rev. 2022 May 25
[6] DeepAMR for predicting co-occurrent resistance of Mycobacterium tuberculosis. Bioinformatics. 2019 Sep 15
[7] A Practical Approach for Predicting Antimicrobial Phenotype Resistance in Staphylococcus aureus Through Machine Learning Analysis of Genome Data. Front Microbiol. 2022 Mar 2
[8] Using Machine Learning To Predict Antimicrobial MICs and Associated Genomic Features for Nontyphoidal Salmonella. J Clin Microbiol. 2019 Jan 30
专家介绍

周华
浙江大学医学院附属第一医院呼吸与危重症医学科副主任,教研室副主任,主任医师,博士生导师;医学博士,美国芝加哥大学访问学者;中华医学会呼吸病学分会青年委员、内科学分会青年委员、呼吸病学分会感染学组委员兼秘书;中国医师协会呼吸系感染工作委员会委员;中国老年医学会呼吸病学分会青年委员;浙江省呼吸系统感染诊治联盟执行主席;浙江省医学会呼吸病学分会青年委员会副主委、感染学组副组长;浙江省医师协会呼吸医师分会感染学组副组长。
本文由《呼吸界》编辑 Jerry 整理,感谢周华老师的审阅修改!
* 文章仅供医疗卫生相关从业者阅读参考
本文完
责编:Jerry


